Recherche translationnelle en santé,
technologie pour la santé et recherche clinique
- > DEVIN
Travaille sur
- > Multimodal MRI-PET data-driven pipeline for the discrimination of prodromal Alzheimer’s Disease
Federico Nemmi, Pierre Payoux, Jérémie Pariente, Patrice Péran
9th ECCN, 2-3 mars 2020, Parisff
- > A whole-brain multimodal discrimination of Parkinsons dDiseas, Multiple System Atrophy and Controls.5th Congress of the European Academy of Neurology, EAN 2019, 29 juin- 2 Juillet 2019 à Oslo, Norvège.
F Nemmi, A Pavy-Le Traon, O Phillips, M Galitzky, W Meissner, O Rascol, P Péran .
- > Discriminating between neurofibromatosis-1 and typically developing children by means of multimodal MRI and multivariate analyses.5th Congress of the European Academy of Neurology, EAN 2019, 29 juin- 2 Juillet 2019 à Oslo, Norvège.
F Nemmi, F Cignetti, C Assaiante, S Maziero, F Audic, P Péran, Y Chaix.
- > A whole-brain multimodal discrimination of Parkinsons dDiseas, Multiple System Atrophy and Controls.Organization for Human Brain Mapping,OHBM 2019, 9-13 juin 2019 à Rome, Italie.
F Nemmi, A Pavy-Le Traon, O Phillips, M Galitzky, W Meissner, O Rascol, P Péran
- > Discriminating neurofibromatosis and healthy children with multimodal MRI and machine learning.Organization for Human Brain Mapping,OHBM 2019, 9-13 juin 2019 à Rome, Italie.
F Nemmi, F Cignetti, C Assaiante, S Maziero, F Audic, P Péran, Y Chaix.
Abstracts rédigés ou co-rédigés
- > 73- Galantamine combined with cognitive rehabilitation on post-stroke cognitive impairment: a proof-of-concept study.Brain Inj. 2024 Oct 3:1-10. doi: 10.1080/02699052.2024.2409355.
Planton M, Nemmi F, Pages B, Albucher JF, Raposo N, Danet L, Péran P, Pariente J.
- > 88- Developmental dyslexia, developmental coordination disorder and comorbidity discrimination using multimodal structural and functional neuroimaging.Cortex. 2022 Dec 17;160:43-54. doi: 10.1016/j.cortex.2022.10.016.
Nemmi F, Cignetti F, Vaugoyeau M, Assaiante C, Chaix Y, Péran P.
- > 56- Insular functional connectivity in migraine with aura.J Headache Pain. 2022 Aug 19;23(1):106. doi: 10.1186/s10194-022-01473-1.
Gollion C, Lerebours F, Nemmi F, Arribarat G, Bonneville F, Larrue V, Péran P.
- > 13- Atypical connectivity in the cortico-striatal network in NF1 children and its relationship with procedural perceptual-motor learning and motor skills.J Neurodev Disord. 2022 Mar 1;14(1):15. doi: 10.1186/s11689-022-09428-y.
Baudou E, Nemmi F, Peran P, Cignetti F, Blais M, Maziero S, Tallet J, Chaix Y.
- > 7- Brain-age estimation accuracy is significantly increased using multishell free-water reconstruction.Hum Brain Mapp. 2022 Feb 10. doi: 10.1002/hbm.25792.
Nemmi F, Levardon M, Péran P.
- > 47- Are morphological and structural MRI characteristics related to specific cognitive impairments in neurofibromatosis type 1 (NF1) children?Eur J Paediatr Neurol. 2020 Jul 31:S1090-3798(20)30144-6. doi: 10.1016/j.ejpn.2020.07.003. Online ahead of print.
Baudou E, Nemmi F, Biotteau M, Maziero S, Assaiante C, Cignetti F, Vaugoyeau M, Audic F, Peran P, Chaix Y.
Voir la publication - > 33- Neuroplasticity and brain reorganization associated with positive outcomes of multidisciplinary rehabilitation in progressive multiple sclerosis: A fMRI study.Mult Scler Relat Disord. 2020 May 6;42:102127. doi: 10.1016/j.msard.2020.102127.
Péran P, Nemmi F, Dutilleul C, Finamore L, Falletta Caravasso C, Troisi E, Iosa M, Sabatini U, Grazia Grasso M.
Voir la publication - > 28- Functional and Structural Integrity of Frontoparietal Connectivity in Traumatic and Anoxic Coma.Crit Care Med. 2020 May 11. doi: 10.1097/CCM.0000000000004406.
Peran P, Malagurski B, Nemmi F, Sarton B, Vinour H, Ferre F, Bounes F, Rousset D, Mrozeck S, Seguin T, Riu B, Minville V, Geeraerts T, Lotterie JA, Deboissezon X, Albucher JF, Fourcade O, Olivot JM, Naccache L, Silva S.
Voir la publication - > 26- Interhemispheric distribution of amyloid and small vessel disease burden in cerebral amyloid angiopathy-related intracerebral hemorrhage.Eur J Neurol. 2020 May 11. doi: 10.1111/ene.14301.
Planton M, Pariente J, Nemmi F, Albucher JF, Calviere L, Viguier A, Olivot JM, Salabert AS, Payoux P, Peran P, Raposo N.
Voir la publication - > 50- Significant Decrease in Hippocampus and Amygdala Mean Diffusivity in Treatment-Resistant Depression Patients Who Respond to Electroconvulsive Therapy.Front Psychiatry. 2019 Sep 19;10:694. doi: 10.3389/fpsyt.2019.00694. Voir la publication
Yrondi A, Nemmi F, Billoux S, Giron A, Sporer M, Taib S, Salles J, Pierre D, Thalamas C, Schmitt L, Péran P, Arbus C.
- > 44- Grey Matter changes in treatment-resistant depression during electroconvulsive therapy.J Affect Disord. 2019 Jul 31;258:42-49. doi: 10.1016/j.jad.2019.07.075.
Yrondi A, Nemmi F, Billoux S, Giron A, Sporer M, Taib S, Salles J, Pierre D, Thalamas C, Rigal E, Danet L, Pariente J, Schmitt L, Arbus C, Péran P.
Voir la publication - > 25- A totally data-driven whole-brain multimodal pipeline for the discrimination of Parkinson’s disease, multiple system atrophy and healthy controlNeuroImage Clinical
Nemmi F, Pavy-Le Traon A, Phillips O.R, Galitzky M, Meissner W.G, Rascol O, Péran P
- > 24- Discriminating between neurofibromatosis-1 and typically developing children by means of multimodal MRI and multivariate analyses2019;1–14
Nemmi F, Cignetti F, Assaiante C, Maziero S, Audic F, Péran P, Chaix Y
Hum Brain Mapp.
- > 21- Discriminating between neurofibromatosis-1 and typically developing children by means of multimodal MRI and multivariate analyses.Hum Brain Mapp. 2019 May 11. doi: 10.1002/hbm.24612.
Nemmi F, Cignetti F, Assaiante C, Maziero S, Audic F, Péran P, Chaix Y.
- > 53- Action and Non-Action Oriented Body Representations: Insight from Behavioural and Grey Matter Modifications in Individuals with Lower Limb Amputation.Biomed Res Int. 2018 Oct 18;2018:1529730. doi: 10.1155/2018/1529730.
Palermo L, Di Vita A, Boccia M, Nemmi F, Brunelli S, Traballesi M, De Giorgi R, Galati G, Guariglia C.
- > 5- MRI supervised and unsupervised classification of Parkinson’s disease and multiple system atrophy.Mov Disord. 2018 Feb 23. doi: 10.1002/mds.27307.
Péran P, Barbagallo G, Nemmi F, Sierra M, Galitzky M, Traon AP, Payoux P, Meissner WG, Rascol O.
- > 39- Multimodal Magnetic Resonance Imaging in Alzheimer’s Disease Patients at Prodromal Stage.J Alzheimers Dis. 2016;50(4):1035-50. doi: 10.3233/JAD-150353.
Eustache P, Nemmi F, Saint-Aubert L, Pariente J, Péran P.
- > 34- Multimodal MRI assessment of nigro-striatal pathway in multiple system atrophy and Parkinson disease.Mov Disord. 2016 Mar;31(3):325-34. doi: 10.1002/mds.26471.
Barbagallo G, Sierra-Peña M, Nemmi F, Traon AP, Meissner WG, Rascol O, Péran P.
- > Parkinson’s disease and local atrophy in subcortical nuclei: insight from shape analysis.Neurobiol Aging. 2015 Jan;36(1):424-33. doi: 10.1016/j.neurobiolaging.2014.07.010.
Nemmi F, Sabatini U, Rascol O, Péran P.
- > A case of logopenic primary progressive aphasia with C9ORF72 expansion and cortical florbetapir binding.J Alzheimers Dis. 2014;42(2):413-20. doi: 10.3233/JAD-140222.
Saint-Aubert L, Sagot C, Wallon D, Hannequin D, Payoux P, Nemmi F, Bezy C, Chauveau N, Campion D, Puel M, Chollet F, Pariente J.
- > Insight on AV-45 binding in white and grey matter from histogram analysis: a study on early Alzheimer’s disease patients and healthy subjects.Eur J Nucl Med Mol Imaging. 2014 Jul;41(7):1408-18. doi: 10.1007/s00259-014-2728-4.
Nemmi F, Saint-Aubert L, Adel D, Salabert AS, Pariente J, Barbeau EJ, Payoux P, Péran P.
- > Comparison between PET template-based method and MRI-based method for cortical quantification of florbetapir (AV-45) uptake in vivo.Eur J Nucl Med Mol Imaging. 2014 May;41(5):836-43. doi: 10.1007/s00259-013-2656-8.
Saint-Aubert L, Nemmi F, Péran P, Barbeau EJ, Payoux P, Chollet F, Pariente J.
Publications rédigées ou co-rédigées
Conjuguant son expertise dans le traitement d’images IRM et TEP avec ses connaissances en statistique et apprentissage automatique Federico Nemmi développe et valide des méthodes innovantes pour l’analyse d’imagerie multimodale visant au diagnostic et au pronostic des maladies neurologiques, neurodégénératives et psychiatriques. Sa recherche se focalise aussi sur le développement d’outils d’analyses qui puissent combiner données d’imagerie et données de sources diverses (ex. échantillons biologiques, données neuropsychologiques) pour améliorer le diagnostic et le pronostic des maladies neurologiques, neurodégénératives et psychiatriques. Les méthodes développés sont librement accessibles sur son GitHub.
Projet
L’imagerie cérébrale multimodale a permis d’observer plusieurs caractéristiques du cerveau, tel que sa morphologie, sa microstructure ou son activité, pour un même sujet. Cette grand quantité d’informations complémentaires est fondamental pour avoir une compréhension holistique de la pathologie ou de la dégénérescence cérébra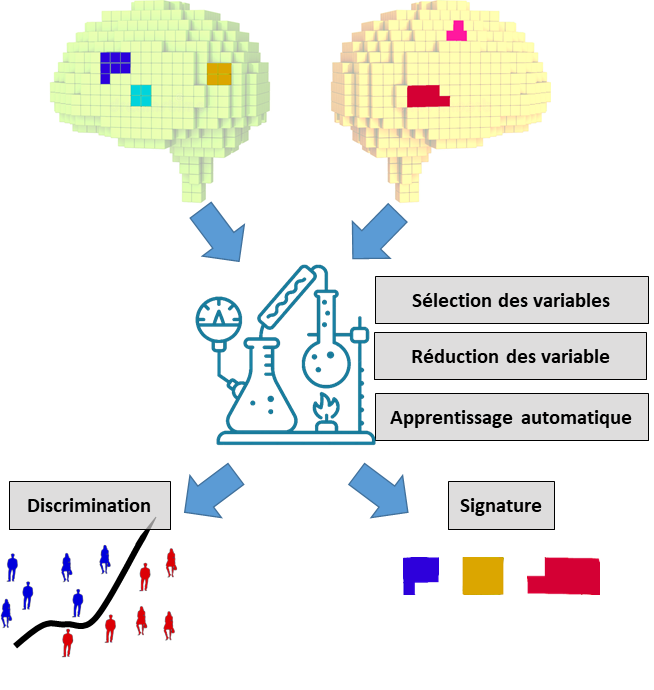 l. Pour mettre à fruit tout leur potentiel, ces informations doivent être analysé avec des méthodes qui puissent prendre en compte leur structure globale et trouver, entre la multiplicité de variables, les éléments les plus pertinents pour distinguer un patient d’un sujet sain, ou pour prédire une échelle symptomatique. J’ai donc développé une pipeline d’analyse que puisse atteindre ces objectifs : à travers l’enchaînement de plusieurs passages de sélection et réduction de variable, couplé avec des modèles issus de l’apprentissage automatique, cette pipeline peut discriminer entre patients et sujet sain tout en mettant en lumière les régions cérébrales et les modalités d’imagerie que plus contribuent à cette discrimination. La pipeline a était validé sur une maladie génétique tel que la neurofibromatose (Nemmi et al., Hum Brain Map, 2019) et sur des syndromes parkinsoniennes (atrophie multi-systématisée, maladie de Parkinson) (Nemmi et al., Neuroim Clin, 2019) avec de données IRM et sur la maladie d’Alzheimer avec de données mixte d’IRM et TEP (European Conference on Clinical Neuroimaging, 2019, présentation orale), en utilisent des jeux de données déjà acquis au sein de ToNIC. De plus, la pipeline est capable de prédire de variable continues, tel que l’âge d’un sujet, à partir de données d’IRM multimodale (Nemmi et al., soumit). Au moment, mes efforts se focalisent sur l’optimisation pour les données TEP. Dans le proche future mon but est d’utiliser la pipeline pour observer la signature multimodale du traitement de la dépression par électroconvulsivothérapie. À terme, j’ envisage d’adapter la pipeline pour inclure des données longitudinale et pour pouvoir exploiter de données tel quel les échelles cliniques ou le tests cognitifs.
l. Pour mettre à fruit tout leur potentiel, ces informations doivent être analysé avec des méthodes qui puissent prendre en compte leur structure globale et trouver, entre la multiplicité de variables, les éléments les plus pertinents pour distinguer un patient d’un sujet sain, ou pour prédire une échelle symptomatique. J’ai donc développé une pipeline d’analyse que puisse atteindre ces objectifs : à travers l’enchaînement de plusieurs passages de sélection et réduction de variable, couplé avec des modèles issus de l’apprentissage automatique, cette pipeline peut discriminer entre patients et sujet sain tout en mettant en lumière les régions cérébrales et les modalités d’imagerie que plus contribuent à cette discrimination. La pipeline a était validé sur une maladie génétique tel que la neurofibromatose (Nemmi et al., Hum Brain Map, 2019) et sur des syndromes parkinsoniennes (atrophie multi-systématisée, maladie de Parkinson) (Nemmi et al., Neuroim Clin, 2019) avec de données IRM et sur la maladie d’Alzheimer avec de données mixte d’IRM et TEP (European Conference on Clinical Neuroimaging, 2019, présentation orale), en utilisent des jeux de données déjà acquis au sein de ToNIC. De plus, la pipeline est capable de prédire de variable continues, tel que l’âge d’un sujet, à partir de données d’IRM multimodale (Nemmi et al., soumit). Au moment, mes efforts se focalisent sur l’optimisation pour les données TEP. Dans le proche future mon but est d’utiliser la pipeline pour observer la signature multimodale du traitement de la dépression par électroconvulsivothérapie. À terme, j’ envisage d’adapter la pipeline pour inclure des données longitudinale et pour pouvoir exploiter de données tel quel les échelles cliniques ou le tests cognitifs.
Travaux antérieurs:
Adaptation de la pipeline pour variables continues – étude sur les meilleurs marqueurs de diffusion de l’âge
S’il est important de pouvoir discriminer entre populations, il est certainement autant important de pouvoir prédire des variables continues tels que des échelles symptomatiques ou la performance à des tests cognitifs. J’ai donc adapté la pipeline pour les variables continues, en retravaillant en particulier les étapes de sélections de variables. J’ai ensuite utilisé la pipeline adaptée pour étudier le diffèrent pouvoir prédictif des paramètres issus de l’IRM de diffusion pour la prédiction de l’âge. J’ai pu mettre en lumière que, malgré tous les paramètres étudiés puissent prédire l’âge, certains paramètres, et notamment ces issues de l’algorithme de reconstruction en eau libre (FW), ont une performance significativement meilleure que les autres.
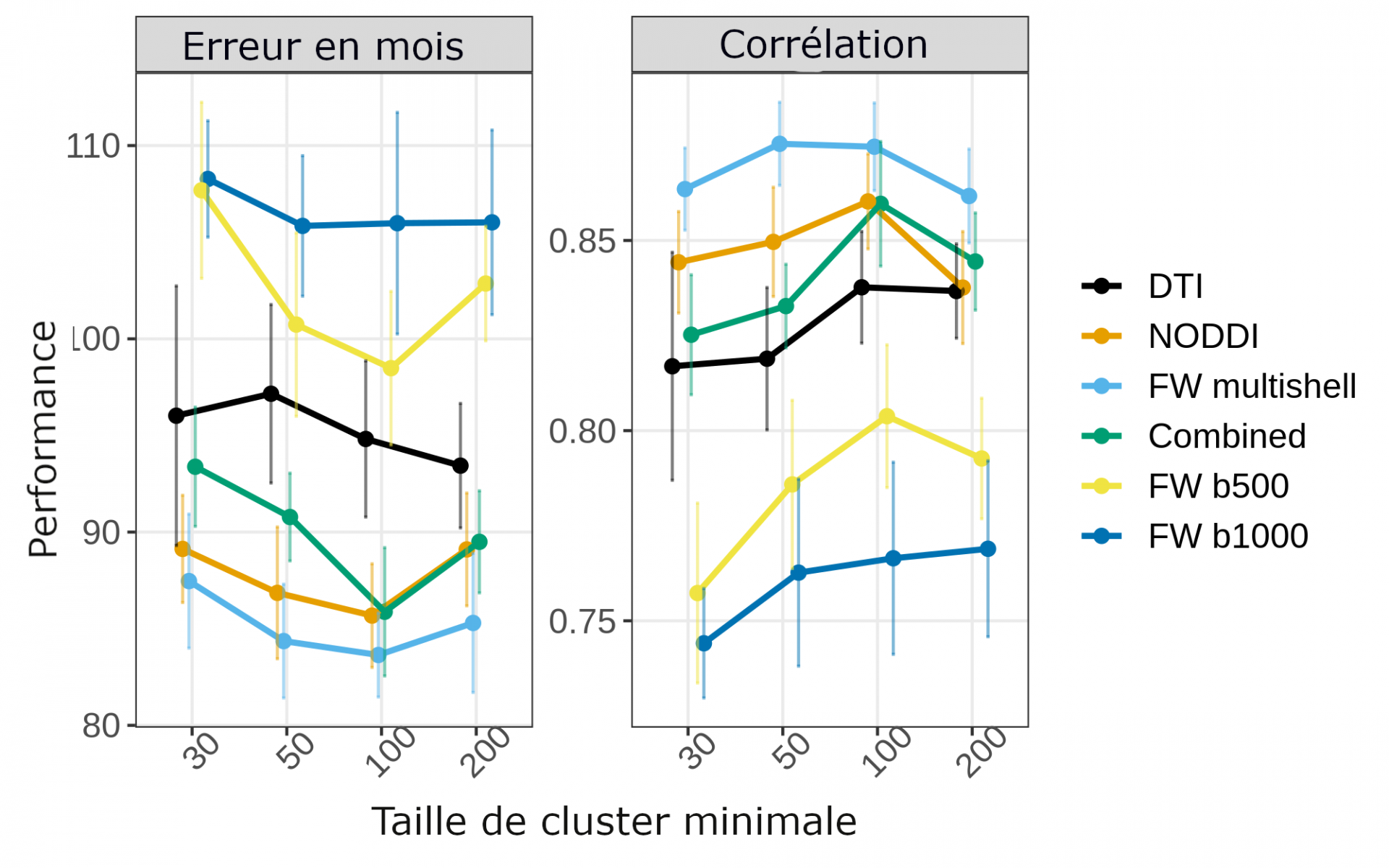
Performances (Erreur en mois = vrai âge – âge prédites ; Corrélation = r de Pearson) pour plusieurs algorithmes de reconstruction de données de diffusion (DTI = tenseur de diffusion ; NODDI = orientation et dispersion d’axones ; FW multishell = eau libre avec plusieurs valeurs de B0 ; Combined = tous les précédents ; FWb500/b100 (eau libre calculé avec une seule valeur de B0).
Analyses multivarié et IRM multimodale pour la discrimination de populations pathologiques
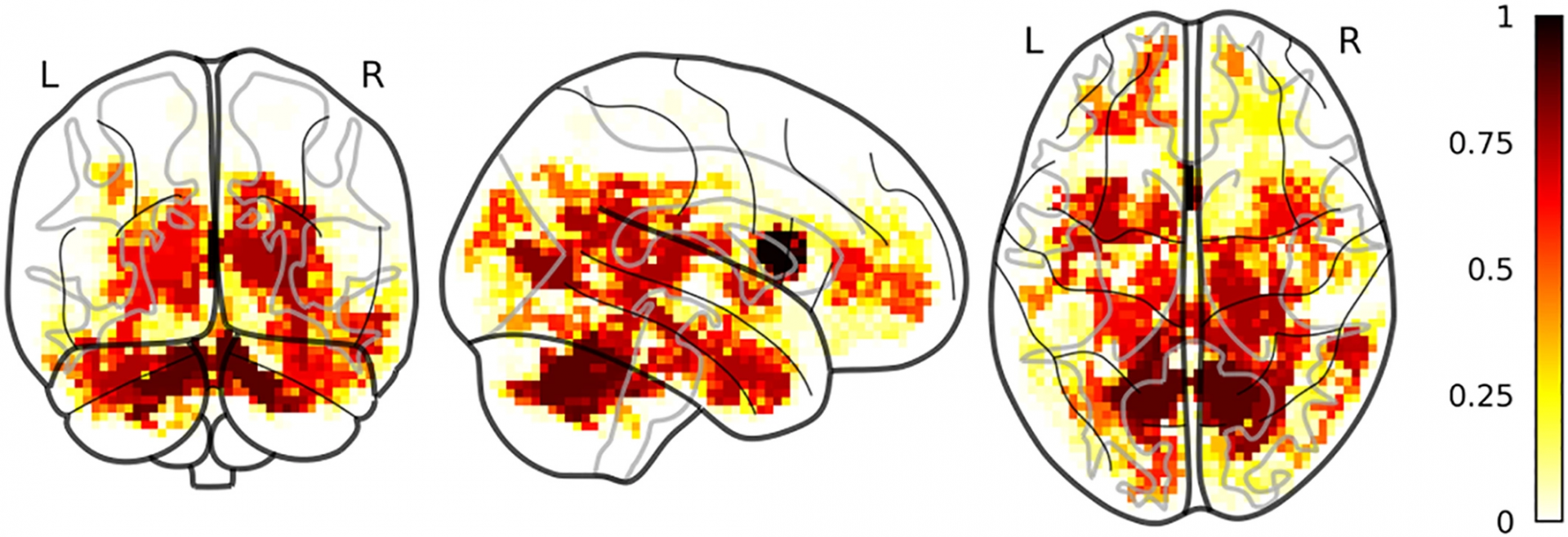
Régions cérébrales plus discriminantes entre enfants sains et atteints de neurofibromatose pour la diffusivité moyenne
Pour pouvoir maximiser l’exploitation des information d’imagerie multimodale, pouvoir trouver la signature multimodale des pathologies cérébrales et discriminer entres populations pathologiques j’ai développé une pipeline d’analyse qui enchaine une série de techniques de sélection et réduction de variables avec une algorithme d’apprentissage automatique. Cette pipeline a était validé sur des pathologies très différentes tels que la neurofibromatose (une pathologie développemental génétique) et la maladie de Parkinson et l’atrophie multi-systématisée.
Pour ce qui concerne la neurofibromatose, j’ai pu discriminer avec enfants atteints de cette pathologie et enfants sain, en montrant que la diffusivité moyenne dans le thalamus et le cervelet est le marqueur plus discriminant (Nemmi et al., Hum Brain Map, 2019).
Ensuite, j’ai pu montrer que ma pipeline peut non seulement discriminer entre sujets sain et malades, mais aussi entre patients atteints de différentes pathologies. Notamment, ma pipeline a était utilisé pour discriminer entre patients avec maladie de Parkinson et patients atteints d’atrophie multi-systématisée et entre ces deux populations et de sujets témoins. Chaque discrimination était associée à une signature multimodale qui était spécifique pour les différentes discrimination (Nemmi et al., Neuroim Clin, 2019).
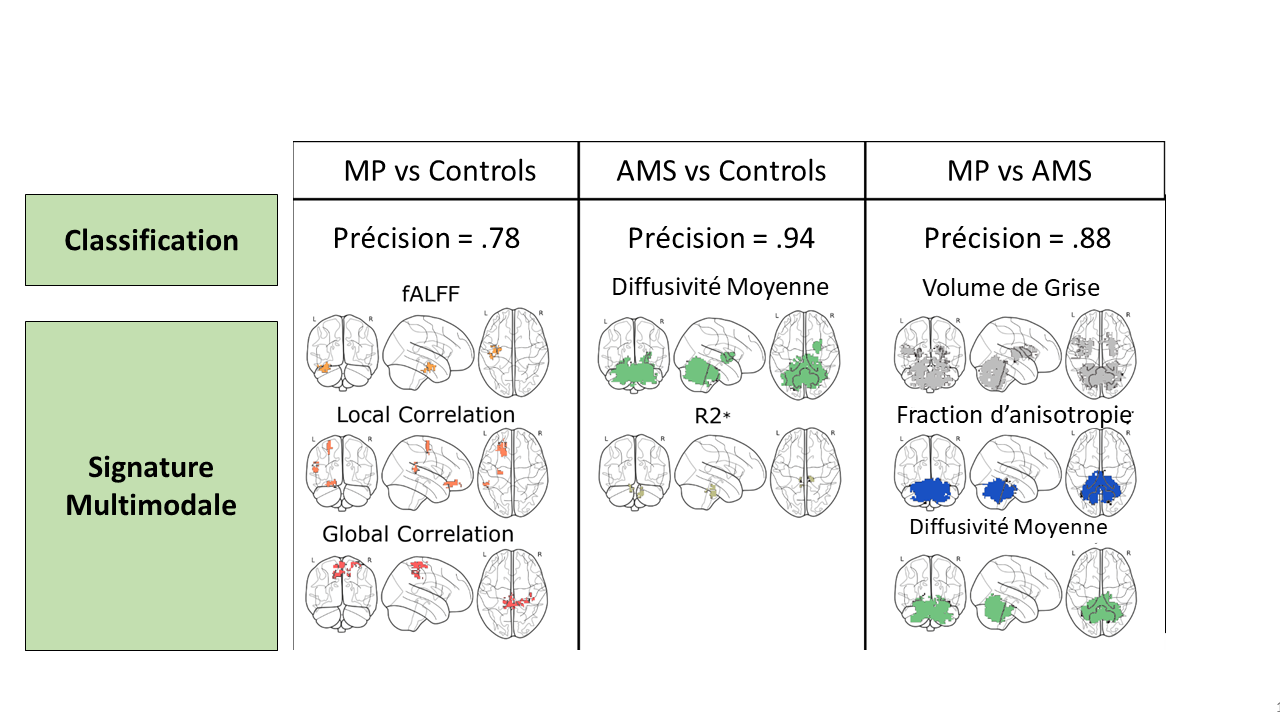
Performances et signatures multimodales pour les différentes discrimination (MP = maladie de Parkinson, AMS = atrophie multi-systématisée)
Association de paramètre d’IRM avec données comportementales et génétique
Pendent mon post-doc au Karolinska Institutet j’ai pu montrer comment l’association entre index d’IRM et données d’autre type (comportementales ou génétiques) améliore la prédiction du comportement future ou de la réponse à un traitement (Nemmi et al., Dev Cogn Neurosci, 2016, 2018)
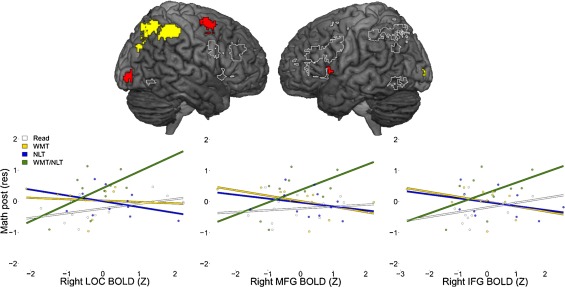
L’activité dans plusieurs régions cérébrales couplé à des mesures cognitives prédis l’efficacité d’un entrainement cognitif pour améliorer les habilités mathématiques
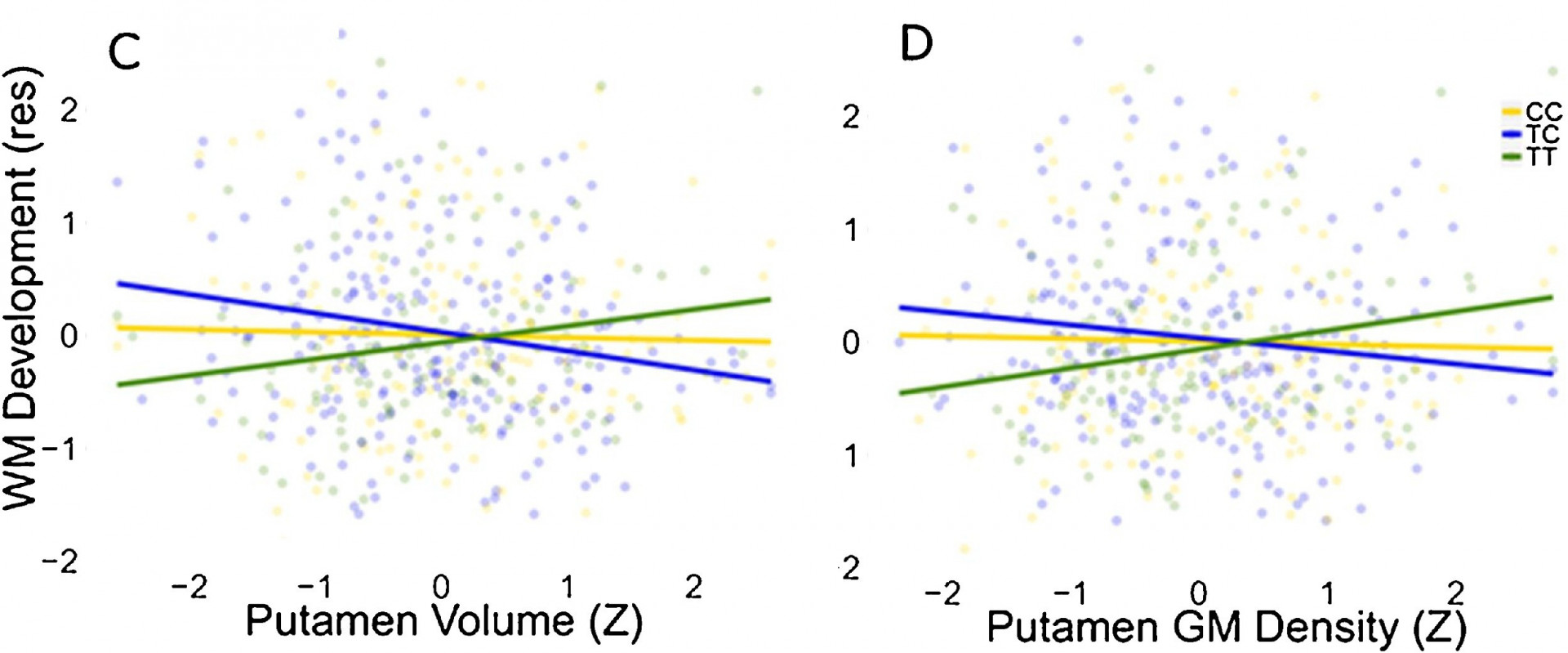
Le volume et la densité de matière grise du putamen prédisent le développement de la mémoire de travail entre 14 et 19 ans seulement en association avec l’information sur un polymorphisme du gène DAT1 (CC, TC ou TT).
Association d’IRM et TEP pour améliorer la compréhension de la pathologie cérébrale
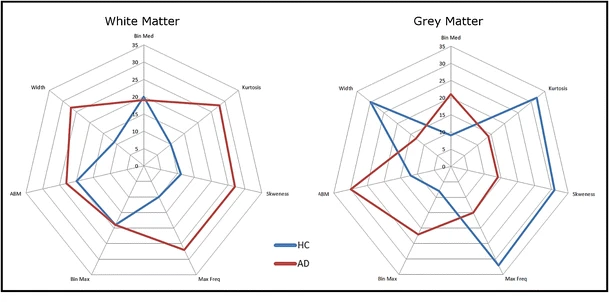
Grace à l’association entre IRM morphologique et TEP amyloïde j’ai pu extraire les histogrammes de charge amyloïde dans la matière blanche et grise des patients Alzheimer et témoins. Une analyse multivariée des index issue des histogrammes de la matière blanche a montré, pour la première fois, que la fixation d’amyloïde dans la matière blanche est en relation avec la pathologie (Nemmi et al., Eur J Nucl Med Mol Imaging, 2014).
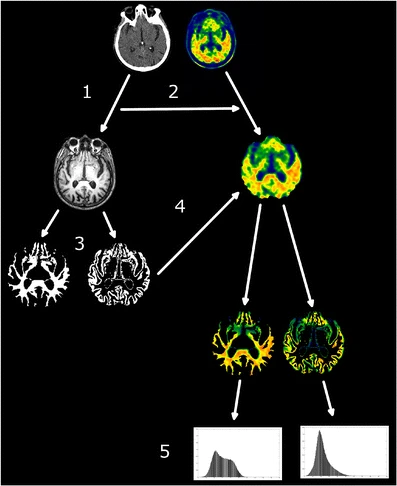
- Pipeline de traitement d’image combinant CT, TEP et IRM
- Différences entre les index issus des histogrammes de la matière blanche (gauche) et grise (droit) entre sujet sain (blu) et patients Alzheimer
L’association d’IRM et TEP a aussi permis de montrer que si une normalisation spatial directe de données de TEP amyloïde sur un template est plus adapté pour l’utilisation clinique, la registration de données TEP sur l’IRM est plus adapté pour la recherche (Saint-Aubert*, Nemmi*, et al., Eur J Nucl Med Mol Imaging , 2014).
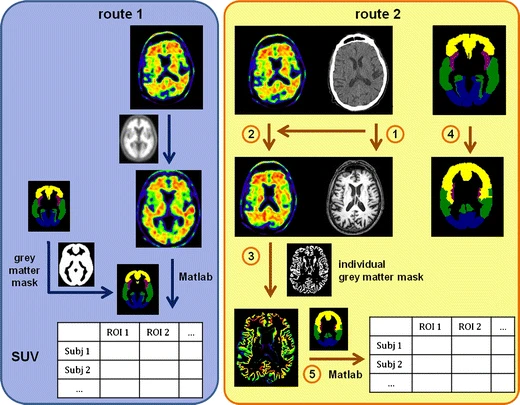
Illustration d’une méthode de registration directe et via le CT scan et l’IRM de l’imagerie PET amyloïde
IRM multiparamètrique et forme de noyaux sous-corticaux
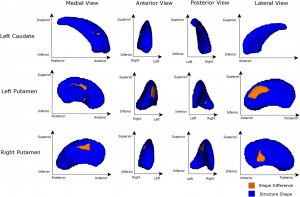
Région des noyaux sous-corticaux (orange) qui mieux différentient entre patients parkinsoniens et témoins
La forme des noyaux sous-corticaux discrimine entre patients Parkinsoniens et sujets témoins mieux que leur volume. De plus, les informations sur la forme mais pas sur le volume de noyaux sous-corticaux corrèle avec la symptomatologie. (Nemmi et al., Neurobiol Aging, 2015).
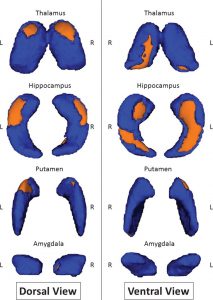
Région des noyaux sous-corticaux (orange) qui mieux différentient entre patients Alzheimer et témoins
Utiliser la forme de noyaux sous-corticaux in association avec l’imagerie de diffusion ou de fer améliore la discrimination par rapport à l’imagerie de diffusion ou de fer seules pour des patients atteints de maladie d’Alzheimer (Eustache, Nemmi et al., J Alzheimer ‘s Dis, 2015)